Ứng dụng của ddPCR trong kiểm nghiệm thực phẩm biến đổi gene (Phần 2)
Đăng bởi Hiep Tran - (Theo Hiep Tran) - 04/05/2022Kiểm nghiệm thực phẩm biến đổi gene sẽ xác nhận và định lượng, nếu có, sinh vật biến đổi gene (GMO) trong các mẫu thực phẩm hoặc thức ăn chăn nuôi của doanh nghiệp, đồng thời thực hiện quy trình xác minh độc lập để giúp doanh nghiệp có thể giao dịch một cách tự tin.
Kiểm nghiệm GMO được thực hiện như thế nào?
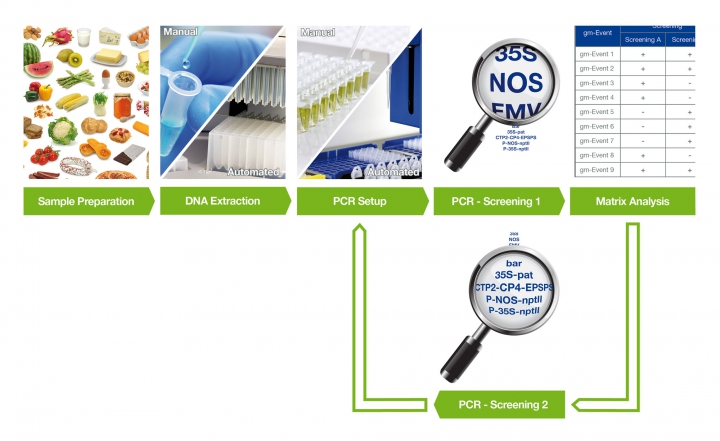
Để xác định GMO, người ta có thể dùng phương pháp ELISA. Phương pháp này dùng để kiểm tra sự có mặt của những loại protein cụ thể dựa trên sự gắn kết đặc hiệu của kháng nguyên và kháng thể. Tuy nhiên, phương pháp ELISA khi được sử dụng để phát hiện GMO có độ nhạy và độ đặc hiệu thấp, lại rất tốn thời gian và công sức. Vì vậy, phương pháp PCR được áp dụng nhiều hơn. Phương pháp này giúp phát hiện những trình tự DNA mới được chèn vào hệ gene của sinh vật. So với ELISA, phương pháp PCR tương đối dễ thực hiện, rẻ, nhạy, đặc hiệu và nhanh hơn.
Việc nghiên cứu các thành phần có nguồn gốc biến đổi gene được thực hiện bằng các phương pháp với các bước liên tục (hoặc đồng thời). Sau khi thu thập mẫu, nucleic acid được chiết ra khỏi phần mẫu thử. Nucleic acid chiết được có thể phải làm sạch tiếp, đồng thời hoặc sau quá trình chiết. Sau đó, các nucleic acid được định lượng, pha loãng và phân tích.
Tuy nhiên, phương pháp Real-time PCR còn gặp nhiều hạn chế
- Real-time PCR cần xây dựng đường chuẩn. Việc này gây cho kĩ thuật viên khá nhiều khó khăn, đặc biệt là những kĩ thuật viên chưa lành nghề.
- Chất ức chế PCR có thể tác động lớn đến độ chính xác của kết quả định lượng
- Real-time PCR phụ thuộc vào cường độ tín hiệu huỳnh quang. Khi tín hiệu huỳnh quang yếu thì kết quả không còn chính xác.
- Gặp giới hạn với hàm lượng DNA nhỏ, khi giá trị Ct trong phản ứng real-time sẽ cao và kết quả không còn đáng tin cậy
- Kết quả định lượng phụ thuộc nhiều vào độ đồng đều của lượng DNA nạp đầu vào và tay nghề kỹ thuật viên
Nếu bạn là người làm trong lĩnh vực công nghệ sinh học, chắc hẳn bạn cũng đã nhiều lần đau đầu với những hạn chế trên của Real-time PCR. Nhưng yên tâm bạn sẽ không còn phải đau đầu thêm nữa khi có sự xuất hiện của Droplet Digital PCR (ddPCR) - tạm dịch là PCR kỹ thuật số vi giọt.

Vai trò và vị trí của ddPCR trong kiểm nghiệm GMO
Về cơ bản, ddPCR cũng bắt đầu với master mix như phương pháp Real-time PCR, cùng với mồi và khuôn DNA. Theo George Karlin Neumann, giám đốc bộ phận khoa học tại trung tâm sinh học kỹ thuật số, công ty sản xuất thiết bị và chẩn đoán y khoa Bio-Rad, “bạn chỉ cần chia master mix thành rất nhiều phần nhỏ, có thể tích bằng nhau (partition)”. Mỗi phần nhỏ này sau đó trở thành một phản ứng độc lập, có hoặc không có tín hiệu huỳnh quang sau khi trải qua các chu kỳ nhiệt, phụ thuộc vào phản ứng có chứa trình tự đích hay không, do đó thuật ngữ digital ra đời.
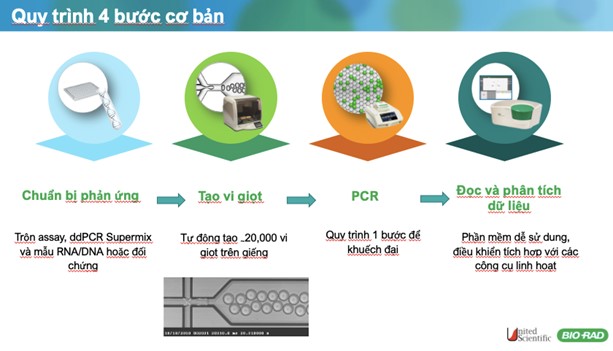
Tính tỷ lệ giữa số phần nhỏ (partition) cho kết quả dương tính và âm tính – sau đó số liệu này được xử lý theo phương pháp thống kê Poisson để xác định nồng độ đích trong dung dịch ban đầu. Kết quả này được xem như tuyệt đối vì không cần dựng đường cong chuẩn Ct (standard Ct curve) như kỹ thuật qPCR.
Digital PCR as an effective tool for GMO quantification in complex matrices: https://www.sciencedirect.com/science/article/pii/S0308814619308271
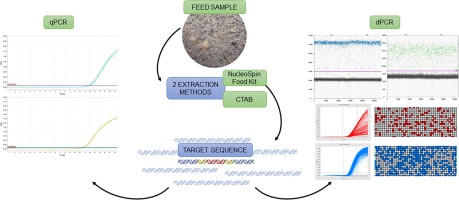
Mặc dù cả qPCR và ddPCR đều là phương pháp định lượng, nhưng trong một số ứng dụng, phương pháp ddPCR lại có ưu thế vượt trội hơn so với qPCR. Trước hết phải kể đến ddPCR có thể phát hiện trình tự đích với hàm lượng ít, theo Huggett, nhà khoa học hàng đầu trong lĩnh vực định lượng nucleic acid tại LGC, Viện đo lường tiêu chuẩn quốc gia Anh trong lĩnh vực đo lường các chỉ tiêu sinh học và hóa học, đây là vấn đề về “độ đặc hiệu” của phương pháp. Ngoài ra, một vấn đề thường gặp với phương pháp dựa vào qPCR là mồi và đầu dò đôi khi có thể gắn vào cả trình tự hoang dại và trình tự đích, do đó không phát hiện các trình tự đích ở hàm lượng dưới 5%. Trong khi đó, “dPCR thường giảm tín hiệu nền từ dạng hoang dại bằng cách pha loãng tất cả DNA, do đó tỷ lệ giữa dạng hoang dại và đột biến thấp hơn nhiều dẫn đến tăng cơ hội phát hiện các đột biến có trong mẫu”.
.png)
Một nghiên cứu công bố năm 2013 của Dany Morisset và cộng sự đã so sánh hiệu quả giữa ddPCR và qPCR trong định lượng GMO ở ngô. Kết quả cho thấy ddPCR có độ chính xác cao hơn với độ chệch (bias) nhỏ hơn. Thêm vào đó, dữ liệu thu được từ ddPCR không bị ảnh hưởng bởi loại mẫu nền.
.png)
Hơn nữa, công suất của ddPCR cũng cao hơn và chi phí rẻ hơn so với qPCR
Tài liệu tham khảo:
Digital PCR as an effective tool for GMO quantification in complex matrices AB Košir, T Demšar, D Štebih, J Žel, M Milavec - Food chemistry, 2019 - Elsevier
The increased use of genetically modified organisms (GMOs) is accompanied by increased complexity of the matrices that contain GMOs. The most common DNA-based approach for GMO detection and quantification is real-time quantitative polymerase chain reaction (qPCR). However, as qPCR is sensitive to inhibitors and relies on standard curves for quantification, it has limited application in GMO quantification for complex matrices. To overcome this hurdle in DNA quantification, we present droplet digital PCR (ddPCR) assays that were designed to target ‘Roundup Ready’ soybean and the soybean reference gene. Three ddPCR assays were transferred from qPCR to QX100/QX200 ddPCR platforms and characterised. Together, the fitness-for-purpose study on four real-life samples and the use of a chamber-based PCR system, showed that dPCR has great potential to improve such measurements in GMO testing and monitoring of food authenticity.
Quantitative analysis of food and feed samples with droplet digital PCR D Morisset, D Štebih, M Milavec, K Gruden, J Žel - PloS one, 2013 - journals.plos.org
In this study, the applicability of droplet digital PCR (ddPCR) for routine analysis in food and feed samples was demonstrated with the quantification of genetically modified organisms (GMOs). Real-time quantitative polymerase chain reaction (qPCR) is currently used for quantitative molecular analysis of the presence of GMOs in products. However, its use is limited for detecting and quantifying very small numbers of DNA targets, as in some complex food and feed matrices. Using ddPCR duplex assay, we have measured the absolute numbers of MON810 transgene and hmg maize reference gene copies in DNA samples. Key performance parameters of the assay were determined. The ddPCR system is shown to offer precise absolute and relative quantification of targets, without the need for calibration curves. The sensitivity (five target DNA copies) of the ddPCR assay compares well with those of individual qPCR assays and of the chamber digital PCR (cdPCR) approach. It offers a dynamic range over four orders of magnitude, greater than that of cdPCR. Moreover, when compared to qPCR, the ddPCR assay showed better repeatability at low target concentrations and a greater tolerance to inhibitors. Finally, ddPCR throughput and cost are advantageous relative to those of qPCR for routine GMO quantification. It is thus concluded that ddPCR technology can be applied for routine quantification of GMOs, or any other domain where quantitative analysis of food and feed samples is needed.
Overview and Recommendations for the Application of Digital PCR: European Network of GMO Laboratories (ENGL) S Pecoraro, G Berben, M Burns, P Corbisier… - 2019 - Publications Office of the European
The so-called digital Polymerase Chain Reaction (dPCR) is a relatively new technique for the detection and quantification of DNA, but its application in analytical laboratories is steadily increasing. In contrast to quantitative real-time PCR, DNA (fragments) can be quantified here without the need for calibration curves. Using dPCR, the PCR mix containing the (target) DNA is partitioned – depending on the device used – currently into a maximum of 10,000,000 small compartments with a volume as low as a few picolitres. These can be either physically distinct compartments on a chip (referred to as chamber-based digital PCR [cdPCR]), or the compartments correspond to water-in-oil droplets (referred to as droplet digital [ddPCR]). Once the PCR has been carried out simultaneously in all compartments/droplets, it is common to both approaches that the number of positive and negative signals for each partition is counted by a fluorescence measurement. With this technique, an absolute quantification of DNA copy numbers can be performed with high precision and trueness, even for very low DNA copy numbers. Furthermore, dPCR is considered less susceptible than qPCR to PCR inhibitory substances that could be co-extracted during DNA extraction from different samples. Digital PCR has already been applied in various fields, for example for the detection and quantification of GMOs, species (animals, plants), human disease bioindicators, food viruses and bacteria including pathogens. When establishing dPCR in a laboratory, different aspects have to be considered. These include, but are not limited to, the adjustment of the type of the PCR master mix used, optimised primer and probe concentrations and the signal separation of positive and negative compartments. This document addresses these and other aspects and provides recommendations for the transfer of existing real-time PCR methods into a dPCR format.
Tóm lại
ddPCR là một công nghệ vô cùng hữu hiệu và vượt trội trong kiểm nghiệm thực phẩm biến đổi gene. Những ưu điểm chính có thể tóm lược như sau:
- Độ chính xác tuyệt đối: ddPCR có thể phát hiện những khác biệt nồng độ rất nhỏ (chỉ 10% pha loãng). Hệ số biến thiên cực nhỏ và khoảng tin cậy (confidence interval) cao.
- Định lượng tuyệt đối mà không cần đường chuẩn.
- Độ lặp lại và độ tuyến tính cao. Kết quả đồng nhất giữa những người thực hiện và những lần thực hiện khác nhau.
- Độ nhạy cao: ddPCR cho phép phát hiện các mục tiêu hiếm trong nền mẫu phức tạp
- Và công nghệ này ít bị ảnh hưởng bởi hiện tượng ức chế, vốn là bài toán nan giải trong các phản ứng PCR thông thường.
Nếu bạn là người đang nghiên cứu về GMO, hãy sử dụng ddPCR để được tối ưu kết quả, tiết kiệm thời gian, công sức và chi phí.