
QC mẫu FFPE bằng hệ thống TapeStation (Agilent) cho hệ thống giải trình tự MGISEQ-2000
Đăng bởi Hải Nam - (Theo Agilent Technologies) - 03/15/2023FFPE là một trong nhiều phương pháp thông dụng để bảo quản mẫu lâm sàng nhờ vào việc duy trì hiệu quả sự nguyên vẹn của mô, phục vụ cho nghiên cứu mô bệnh học và khảo sát hình thái. Tuy nhiên, chất lượng DNA tách từ mẫu FFPE thường bị biến động do liên kết bắt nối của formaldehyde và sự phân huỷ nucleic acid. Vì vậy, việc đánh giá chất lượng mẫu FFPE là cực kỳ quan trọng đối với các ứng dụng có độ nhạy và chi phí cao như giải trình tự. TapeStation 4150 (Agilent) hỗ trợ đánh giá khách quan độ biến chất của mẫu DNA FFPE nhờ vào Chỉ số nguyên vẹn DNA (DIN) và tự động tính toán phổ kích thước, nồng độ của các nhóm phân đoạn. Quy trình chuẩn bị thư viện có thể được điều chỉnh và tối ưu kịp thời dựa trên dữ liệu chất lượng, kích thước thu nhận từ TapeStation 4150 để đảm bảo thư viện cuối có chất lượng đáp ứng các yêu cầu đề ra.
Tóm tắt
Cố định formalin và vùi parafiin (FFPE) là một trong nhiều phương pháp thông dụng để bảo quản mẫu lâm sàng và nghiên cứu y học chuyển giao. Để đảm bảo giải trình tự thành công DNA tách từ mẫu FFPE với độ chính xác cao, cần phải thực hiện bước kiểm tra chất lượng (QC) mẫu DNA và mẫu phân đoạn đầu vào tại những bước quan trọng trong quy trình tạo thư viện giải trình tự NGS. Hệ thống TapeStation 4150 (Agilent) được dùng để thực hiện QC cho toàn bộ quy trình tạo thư viện từ năm mẫu DNA FFPE trên hệ thống giải trình tự MGISEQ-2000RS (MGI Tech). Kết quả giải trình tự thu được có chất lượng cao nhờ quá trình QC nhiều lần xuyên suốt quy trình chuẩn bị thư viện: tại bước chuẩn bị mẫu ban đầu, bước tiền và hậu PCR, đánh giá thư viện cuối.
Thí nghiệm
Vật liệu
Một mẫu FFPE tham chiếu do GeneWell cung cấp. Các mảnh khối u FFPE từ bệnh nhân ung thư tuyến giáp, ung thư cổ tử cung, ung thư đại trực tàng và ung thư vú được lấy từ những người cho giấu tên. DNA được tách chiết bằng máy tách tự động MGISP-NE32 và bộ kit MGIEasy FFPE Genomic DNA Extraction Prepacked (MGI Tech). Bước phân đoạn mẫu DNA bằng sóng siêu âm được thực hiện bằng thiết bị xử lý siêu âm tập trung ME220 (Covaris, p/n 500444). Tạo thư viện bằng kit MGIEasy Universal DNA Library Prep. Kit SureSelect XT HS2 DNA Target Enrichment (Agilent, p/n G9987A) được dùng làm mẫu dò và hoá chất lai để làm giàu mục tiêu NGS. Thiết bị TapeStation 4150 (Agilent) được bố trí làm công cụ phân tích kiểm tra chất lượng DNA, sử dụng Agilent Genomic DNA ScreenTape assay (p/n 5067-5365) và hoá chất hỗ trợ (p/n 5067-5366), Agilent D1000 ScreenTape assay (p/n 5067-5582) và hoá chất hỗ trợ (p/n 5067-5583), Agilent High Sensitivity D1000 ScreenTape assay (p/n 5067-5584) và hoá chất hỗ trợ (p/n 5067-5585).
Tách chiết DNA
Tách chiết nucleic acid từ tất cả các mẫu bằng máy MGISP-NE32 và kit MGIEasy FFPE Genomic DNA Extraction Prepacked theo quy trình chuẩn của nhà sản xuất. Độ nguyên vẹn và nồng độ của mẫu DNA bộ gene (gDNA) được xác định bằng hệ thống TapeStation 4150, sử dụng Genomic DNA ScreenTape và hoá chất hỗ trợ tương ứng, theo quy trình chuẩn của nhà sản xuất.
Chuẩn bị thư viện
Sử dụng kit SureSelect Human All Exon V8 kết hợp với kit MGIEasy Universal DNA Library Prep để chuẩn bị thư viện. D1000 ScreenTape và High Sensitivity D1000 ScreenTape lần lượt được sử dụng với hoá chất hỗ trợ phù hợp để QC thư viện tiền PCR và hậu PCR. Nồng độ và khối lượng thư viện được đo bằng Qubit 4.0 và kit Qubit dsDNA HS Assay (Thermo Fisher). Kích thước các vệt mục tiêu được phân tích bằng TapeStation theo quy trình thí nghiệm chuẩn của nhà sản xuất.
Giải trình tự toàn bộ exome
Sau khi PCR, trộn 60 ng từ mỗi mẫu (tổng cộng bốn mẫu) và mẫu chuẩn FFPE thành một mẫu 300 ng. Sản phẩm PCR đã trộn được biến tính bằng nhiệt rồi gắn khép vòng bằng một phân tử nẹp oligo, tạo thành DNA vòng mạch đơn (ssCir DNA) đóng vai trò thư viện cuối. ssCir DNA được nhân bản để tạo thành một nanoball DNA (DNB) với số lượng bản sao 300 đến 500 trong một phân tử. Các DNB được nạp vào nanoarray, tiến hành đọc 100 base pair-end read và giải trải trình tự bằng phương thức Probe-Anchor Synthesis tổ hợp (cPAS) trên hệ MGISEQ-2000. Dữ liệu giải trình tự được đánh giá theo hai tiêu chí: lượng dữ liệu thu được có vượt 500 M read và chất lượng giải trình tự (Q30) có lớn hơn 80% không.
Kết quả và biện luận
Chất lượng gDNA thu từ mẫu FFPE
Quy trình tách chiết nucleic acid FFPE gồm các bước giải paraffin, rửa và dung giải. Bước giải paraffin có tác động rất quan trọng đến độ nguyên vẹn của DNA. Nếu bước giải paraffin quá lâu có thể dẫn tới nucleic acid bị phân huỷ, nhưng nếu quá ngắn hoặc có các thao tác giải paraffin không hiệu quả thì hiệu suất tách chiết có thể bị giảm và ảnh hưởng đến độ nguyên vẹn DNA.
Hệ tách chiết MGISP-NE32 có thể thu hồi đủ lượng gDNA cần cho NGS. Nồng độ các phân đoạn DNA được đo bằng Qubit. Chất lượng DNA (DIN) và kích thước của các phân đoạn chính được phân tích bằng TapeStation (Bảng 1 và hình 1). Một số mẫu có sự phân huỷ nặng, được thể hiện qua chỉ số DIN thấp.
Bảng 1. DNA từ mẫu FFPE được phân tích chất lượng (DIN) và kích thước (bp) bằng hệ thống TapeStation 4150 (Agilent). Nồng độ (ng/μl) được xác định bằng Qubit. Kích thước các phân đoạn (đỉnh) chính được xác định bằng phần mềm TapeStation, tương ứng với đỉnh cao nhất được thể hiện trong điện di đồ.
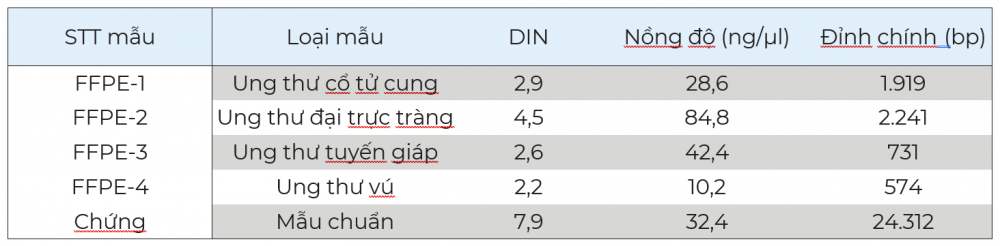
Hình 1. Phân tích gDNA tinh sạch từ mẫu FFPE bằng hệ thống TapeStation 4150, sử dụng Agilent Genomic DNA ScreenTape assay và hoá chất hỗ trợ. Bên trái là hình ảnh mô phỏng miếng gel và bên phải là điện di đồ. A1 là thang được cung cấp kèm theo bộ kit gDNA (100 bp-48,5 kb), B1-E1 tương ứng với bốn mẫu FFPE 1-4 và H1 là mẫu DNA FFPE chuẩn.
Chất lượng thư viện trước tiền lai bắt (pre-capture)
Mẫu gDNA FFPE được dùng để chuẩn bị thư viện, bắt đầu với việc phân cắt thành các đoạn có kích thước gần bằng nhau bằng máy xử lý siêu âm Covaris. Phương pháp phân cắt được tối ưu hoá trước dựa trên kích thước mẫu đo được từ TapeStation. Khoảng 400 ng của mỗi mẫu được đem đi phân đoạn bằng siêu âm. Thời gian phân cắt cho mỗi mẫu sẽ khác nhau tuỳ theo kích thước. Mẫu gDNA lớn hơn 13 kb sẽ được xử lý trong 155 giây, mẫu nhỏ hơn 13 kb sẽ xử lý trong 125 giây. Mọi thông số phân cắt khác được cài giống nhau không phụ thuộc kích thước như sau: công suất đến tối đa 75 W, hệ số sử dụng 20%, 1000 chu kỳ phóng.
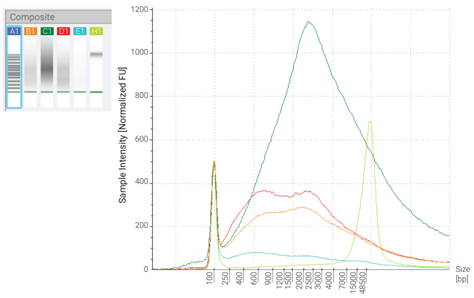
Thư viện được tạo ra từ mỗi mẫu phân đoạn, sử dụng kit MGIEasy Universal DNA Library Prep. Để đảm bảo tính đồng nhất, mỗi thư viện được thực hiện 12 chu kỳ PCR. Thư viện tiền lai được phân tích phổ kích thước phân đoạn và nồng độ bằng D1000 ScreenTape và hoá chất hỗ trợ (Bảng 2). Kết quả cho thấy tất cả các mẫu đều có các đỉnh kích thước đồng đều trong khoảng 200-300 bp (Hình 2).
Bảng 2. Lượng DNA phân cắt bằng siêu âm được dùng để tạo thư viện tiền PCR và nồng độ, khối lượng, kích thước phân đoạn của thư viện tiền PCR. Nồng độ và khối lượng được xác định bằng Qubit. Kích thước của đỉnh chính trong thư viện pre-capture được xác định bằng TapeStation 4150 và là đỉnh cao nhất.
Hình 2. Phân tích thư viện tiền lai bắt bằng hệ thống TapeStation 4150, D1000 ScreenTape và hoá chất hỗ trợ. Bên trái là hình ảnh mô phỏng miếng gel và bên phải là điện di đồ. Đỉnh chính của mỗi thư viện tương ứng với điểm cao nhất và rơi vào khoảng 300 bp ở mọi mẫu, các phân đoạn tập trung chủ yếu trong khoảng 200-500 bp.
Chất lượng của thư viện cuối
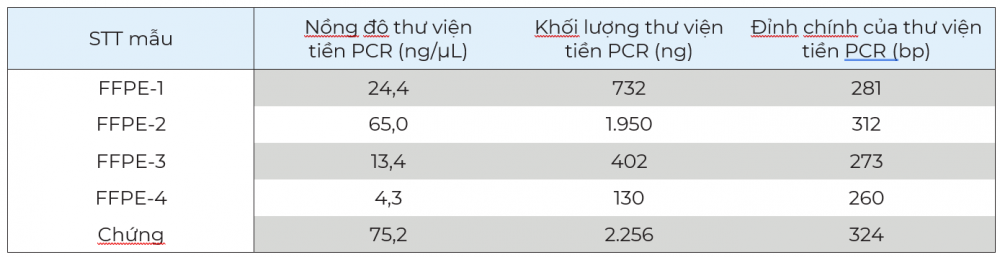
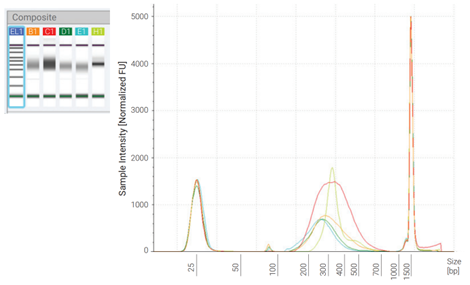
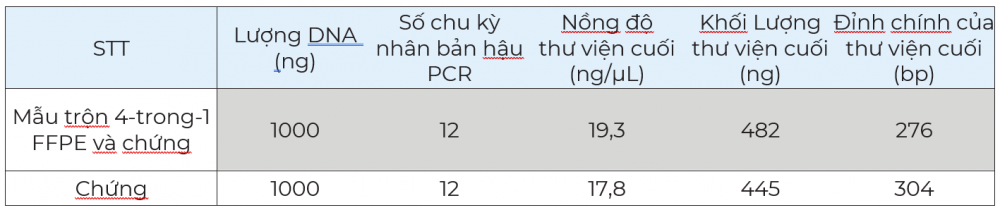
Mẫu tiền lai bắt được trộn lại để tạo thành một thư viện dùng cho bắt lai hoá (hybridization capture) và giải trình tự. Các mẫu tiền lai được trộn theo công thức 300 ng:300 ng:300 ng:100 ng (lần lượt các mẫu 1-4). Mẫu tiền lai FFPE-4 có nồng độ thấp hơn các mẫu khác, do đó cần dùng lượng ít hơn khi trộn. Tổng lượng thư viện để bắt lai hoá là 1.000 ng. Một thư viện khác được chuẩn bị từ 1.000 ng mẫu chuẩn. Bước nhân bản 12 chu kỳ PCR được thực hiện cho mẫu trộn và thư viện chuẩn để tạo ra thư viện cuối. Bước bắt lai hoá được thực hiện bằng SureSelect Human All Exon V8 và kit SureSelect XT HS2 DNA. Bước QC thư viện cuối được tiến hành bằng TapeStation, HS D1000 ScreenTape và hoá chất hỗ trợ. Kích thước của thư viện cuối rải trong khoảng 200-500 bp, tổng lượng là hơn 300 ng, đạt yêu cầu để giải trình tự trên hệ MGI (Bảng 3 và hình 3).
Bảng 3. Lượng thư viện tiền lai sử dụng cho bước bắt lai hoá, sau nhân bản làm giàu và nồng độ, khối lượng thư viện cuối. Khối lượng và nồng độ DNA được xác định bằng Qubit. Kích thước (bp) của thư viện cuối được xác định bằng hệ TapeStation 4150, High Sensitivity D1000 ScreenTape assay và hoá chất hỗ trợ.
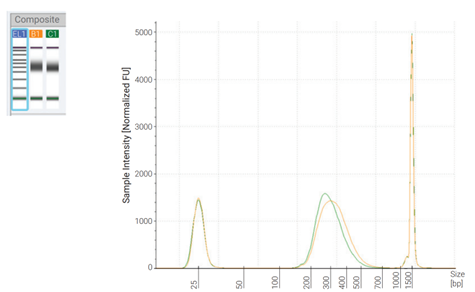
Hình 3. Chất lượng của thư viện cuối được phân tích bằng hệ TapeStation 4150. B1 là mẫu chuẩn, C1 là mẫu trộn FFPE.
Chất lượng của kết quả giải trình tự
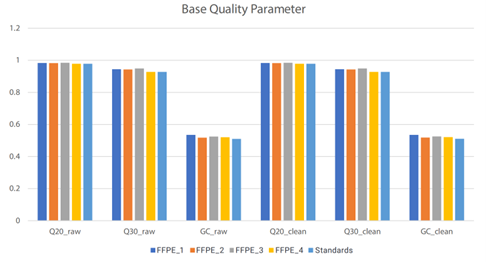
Thư viện cuối được giải trình tự trên hệ MGISEQ-2000RS, sử dụng kit FCS PE100 Rapid Sequencing. Mỗi dữ liệu mẫu được chuẩn hoá thành 7,5 Gb để phân tích. Mọi dữ liệu đều đạt chỉ tiêu nghiệm thu: lượng giải trình tự vượt chuẩn 550 M read trên MGISEQ-2000 và chất lượng giải trình tự (Q30) lớn hơn 90% trước và sau lọc (Hình 4). Kết quả này cho thấy chất lượng mẫu tách chiết và độ đồng nhất, độ phức tạp của thư viện giải trình tự thu được từ bước làm giàu mục tiêu là tối ưu.
Hình 4. Các thông số chất lượng giải trình tự cho bốn mẫu FFPE và mẫu chuẩn.
Kết luận
Trong quy trình chuẩn bị mẫu giải trình tự sử dụng hệ thống MGI, hệ TapeStation 4150 có thể được dùng để QC mẫu tại nhiều giai đoạn. Trước tiên, TapeStation được dùng để phân tích phổ kích thước gDNA tách từ mẫu FFPE. Những kết quả này được dùng để xác định các thông số phân mảnh của mẫu. Thứ hai, hệ TapeStation được dùng sau bước phân đoạn để xác nhận phổ kích thước của mẫu đã phân cắt. Thứ ba, trong bước tạo thư viện, hệ TapeStation có thể đánh giá phổ kích thước của thư viện tiền lai bắt. Thứ tư, phân tích thư viện cuối đã làm giàu mục tiêu bằng TapeStation để đảm bảo kích thước của thư viện đạt yêu cầu của hệ thống giải trình tự. QC bằng hệ thống TapeStation đã hỗ trợ để thu được kết quả giải trình tự thành công cho thí nghiệm.




